比较基因组分析:构建带有分化时间的物种进化树
1 准备蛋白质序列输入文件
1.1 激活环境
xxxxxxxxxx11conda activate comp_genome1.2 安装软件
Gblocks、muscle、modeltest、raxml-ng、PAML、mcmctree
11conda install <software>1.3 多序列比对和保守区域提取:muscle和Gblocks
输入文件:Orthofinder的运行结果文件夹中Single_Copy_Orthologue_Sequences的112条单拷贝基因
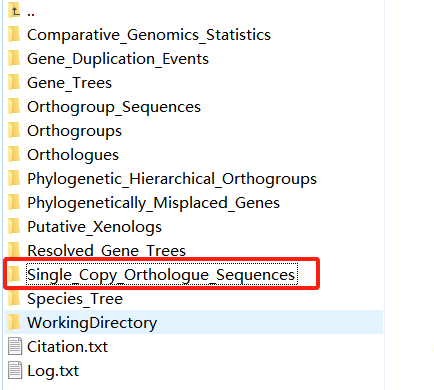
新建muscle_And_gblocks.sh运行文件,先进行多序列比对获得保守结构域,再将保守结构域进行提取。
101cd /home/hztext/dai/Comp_Genome/Nine_ortho_result/Single_Copy_Orthologue_Sequences/2for i in *.fa3do4 file=${i%.fa*}5 muscle -in "${file}.fa" -out "${file}.aln" ## 多序列比对6 Gblocks "${file}.aln" -b4=5 -b5=h -t=p -e=.gb ## 提取保守序列7 seqkit seq "${file}.aln.gb" -w 0 > "${file}.new.aln" ## 多行序列并为一行8 awk '{if (NR%2==1) print substr ($1, 1, 4); else print $0}' "${file}.new.aln" > "${file}.final.aln" ## ID 修饰9done10bash /home/hztext/dai/Comp_Genome/Demo_deal/reminder.sh "您的多序列比对等结果已输出"1.4 串联蛋白质序列
1cd /home/hztext/dai/Comp_Genome/Nine_ortho_result/Single_Copy_Orthologue_Sequences/2#串联文件3seqkit concat *.final.aln > all.fa4#并去除文件中的空格(因为不同aa之间存在空格,像ncbi的格式)5sed -i "s/\ //g" all.fa6bash /home/hztext/dai/Comp_Genome/Demo_deal/reminder.sh "您的单拷贝序列已串联完毕"final.aln文件:
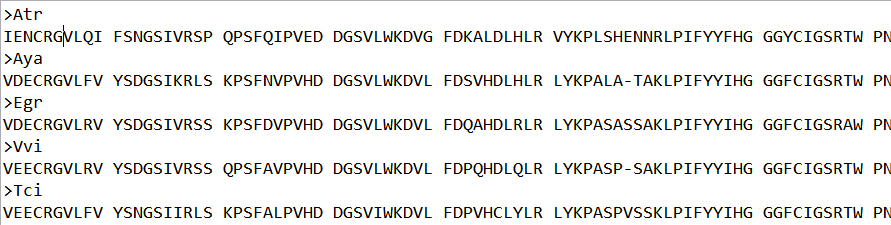
all.fa文件:将串联文件中的序列ID改为物种名简写,检查每个物种的氨基酸数量是否一致(必须一致)。

1.5 fa格式转换为phy格式
新建trans_fa_To_phy.py命令文件。
111import re2with open('all.fa', 'r') as fin:3 sequences = [4 (m.group(1), ''.join(m.group(2).split()))5 for m in re.finditer(r'(?m)^>([^\n]+)[^\n]*([^>]*)', fin.read())6 ]7with open('all.phy', 'w') as fout:8 fout.write('%d %d\n' % (len(sequences), len(sequences[0][1])))9 for item in sequences:10 fout.write('%-20s %s\n' % item)11
新建do_trans_fa_To_phy.sh命令文件执行trans_fa_To_phy.py程序。
41cd /home/hztext/dai/Comp_Genome/Nine_ortho_result/Single_Copy_Orthologue_Sequences/2python /home/hztext/dai/Comp_Genome/Demo_deal/trans_fa_To_phy.py3bash /home/hztext/dai/Comp_Genome/Demo_deal/reminder.sh "您转phy格式已完成"4
格式转化结束后,在每个物种的名后添加8个空格

2 物种进化树构建
2.1 利用modeltest-ng预测最佳的模型
新建modeltest.sh文件并执行
31cd /home/hztext/dai/Comp_Genome/Nine_ortho_result/Single_Copy_Orthologue_Sequences/2modeltest-ng -i all.fa -d aa -o modeltest -p 453bash /home/hztext/dai/Comp_Genome/Demo_deal/reminder.sh "您的建树模型预测已完成"-i:蛋白质序列文件→包含所有物种比对后的保守结构域串联文件
-d:aa代表输入的文件是氨基酸序列(amino acid)
-o:输出文件前缀
-p:线程数

modeltest.out文件中的BIC(贝叶斯信息准则)最佳模型
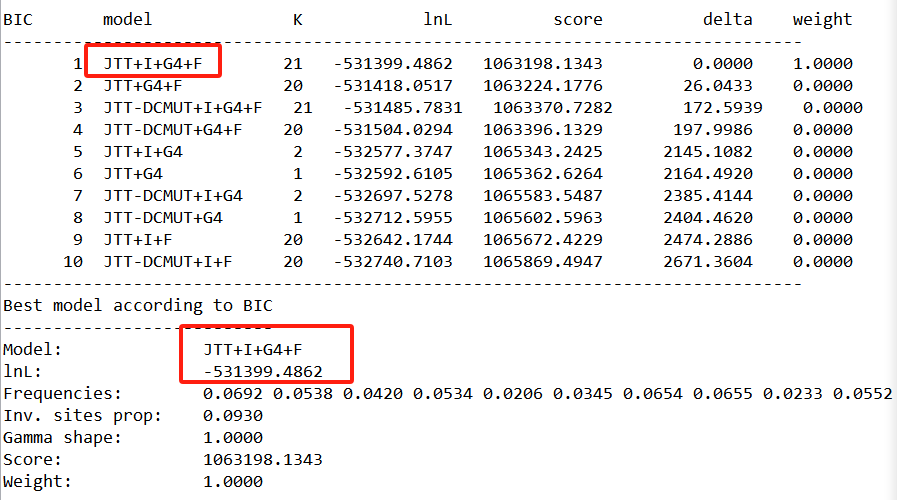
2.2 利用raxml-ng来构建物种进化树
31cd /home/hztext/dai/Comp_Genome/Nine_ortho_result/Single_Copy_Orthologue_Sequences/2raxml-ng --all --msa all.fa --model JTT+I+G4+F --bs-trees 1003bash /home/hztext/dai/Comp_Genome/Demo_deal/reminder.sh "您的raxml-ng建树已完成"–msa:输入蛋白质序列文件;
–model:最佳建树模型。
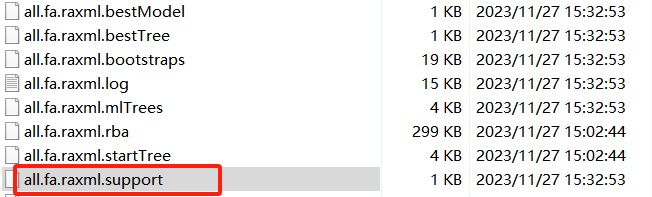
all.fa.raxml.support是构建的物种进化树文件,如下
xxxxxxxxxx11(((Cma:0.008612,Ccl:0.007167)100:0.099212,((TLG:0.012426,Tci:0.010765)100:0.005563,Tsi:0.020005)100:0.063715)100:0.038323,((Atr:0.575439,Vvi:0.159512)100:0.033988,Egr:0.287300)100:0.074467,Aya:0.132865);
3 构建带有进化时间的物种进化树
3.1 利用PAML估计物种的进化时间
基于已知物种间的分化时间,估计其余物种的进化时间,可以从已发表的基因组文章、TimeTree网站获取。
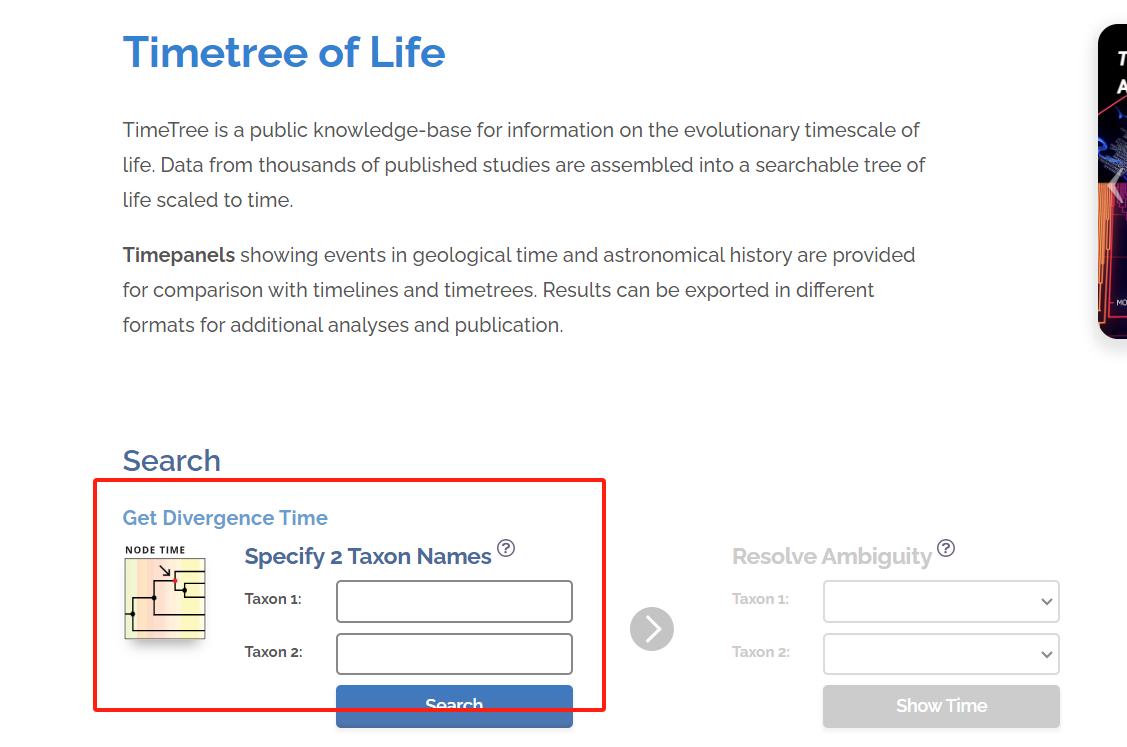
3.2 物种进化树输入文件预处理
下载all.fa.raxml.support文件(无根树)到windows,使用iTOL网站查看进化树,修改为有根树并指定根分支(外类群中进化最早的)。
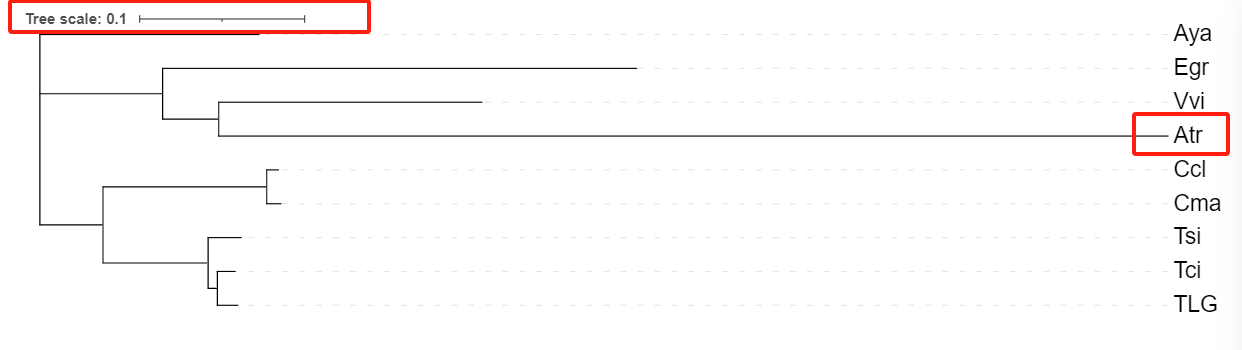
根据进化树中分支的长度,可以发现Atr的分支长度最长,即相比于其他物种的进化时间更早。
为了后续进化树的展示效果,我们将Atr分支设置为根分支(root),并忽略分支长度。
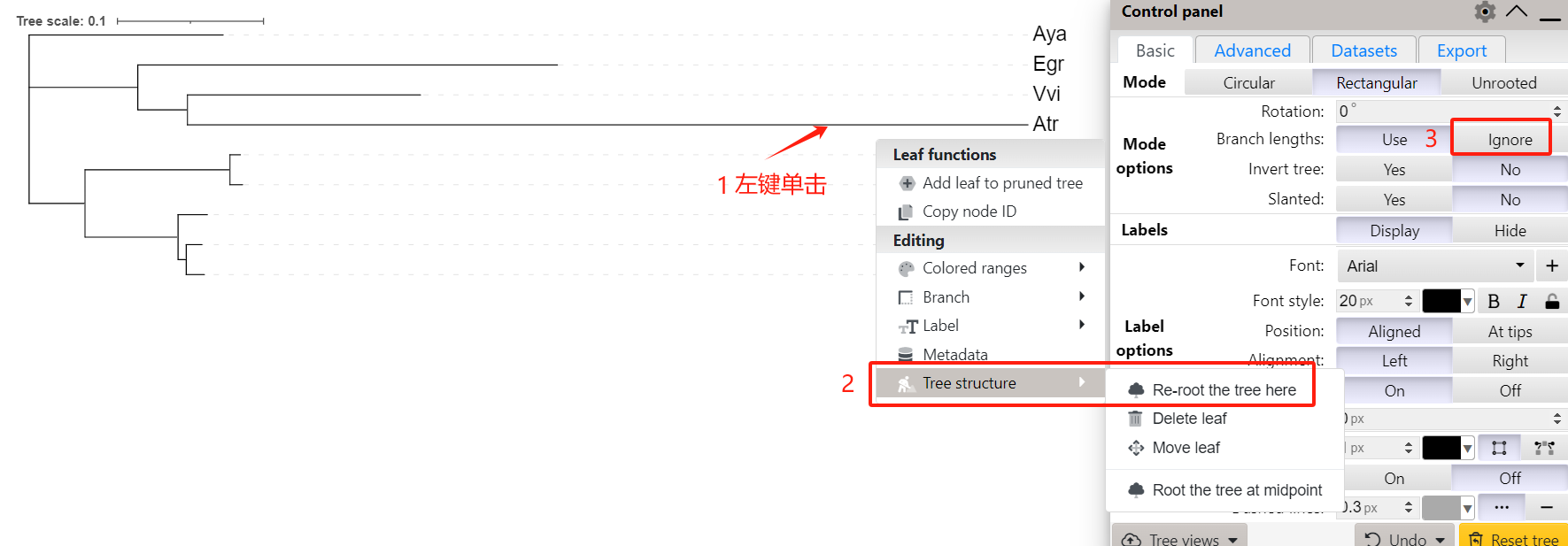
导出修改后的进化树文件,上传至服务器DiffTimetree文件夹。
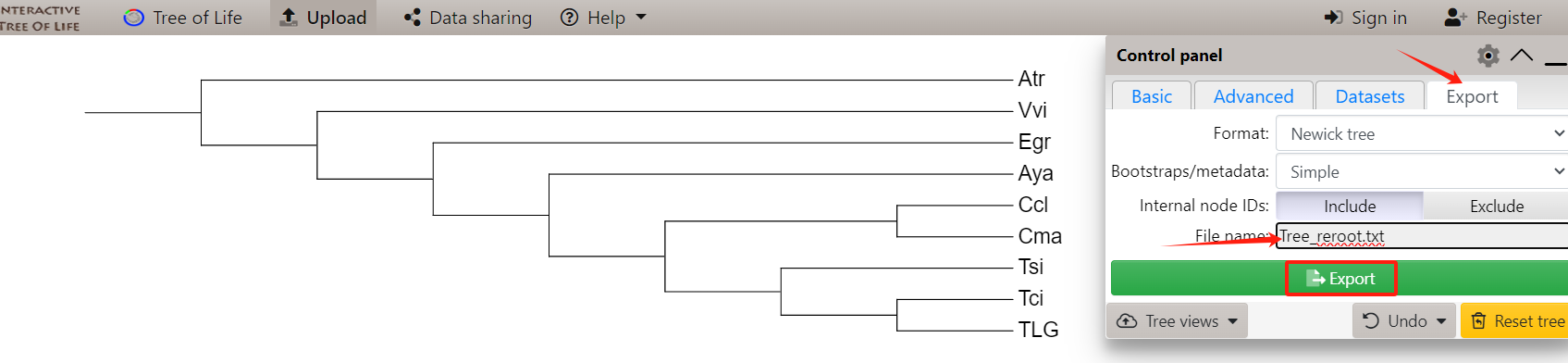
新建tree_deal.r命令文件。
树文件:
x1(Atr:0.2877195,(Vvi:0.159512,(Egr:0.287300,(Aya:0.132865,((Ccl:0.007167,Cma:0.008612)100:0.099212,(Tsi:0.020005,(Tci:0.010765,TLG:0.012426)100:0.005563)100:0.063715)100:0.038323)100:0.074467)100:0.033988):0.2877195);
tree_deal.r:
61if(!requireNamespace("ape", quietly = TRUE)) install.packages("ape")2library(ape)3MLtree <- read.tree("Tree_reroot.txt") #读取单拷贝进化树4MLtree$edge.length <- NULL #删除分支长度信息5MLtree$node.label <- NULL6write.tree(MLtree,"Tree_reroot.tree") #输出只有物种名的进化树新建do_tree_deal.sh命令文件执行tree_deal.r,处理进化树文件。
21cd /home/hztext/dai/Comp_Genome/Nine_ortho_result/DiffTimetree/2Rscript /home/hztext/dai/Comp_Genome/Demo_deal/tree_deal.r修改上述进化树,增加物种分化时间(只添加确定的2-3个),转为以下格式的进化树输入文件:
x19 12(Atr,(Vvi,(Egr,(Aya,((Ccl,Cma)'>1.5<5.7',(Tsi,(Tci,TLG))))))'>179.9<205.0');
9代表树种数量,1固定不变。
3.3 利用PAML的mcmctree构建带有分化时间的进化树
准备输入文件all.phy、Tree_reroot.tree、mcmctree.ctl
mcmctree.ctl:(修改seqfile、 treefile的文件位置,usedata设为3)
x1seed = -12seqfile = ./all.phy3treefile = ./Tree_reroot.tree4outfile = out5ndata = 1 ## 必须注意为 17seqtype = 2 * 0: nucleotides; 1:codons; 2:AAs ## 序列格式,蛋白就选择 AA8usedata = 3 * 0: no data; 1:seq like; 2:use in.BV; 3: out.BV ## 此步骤先设为 39clock = 3 * 1: global clock; 2: independent rates; 3: correlated rates10RootAge = <450.0 * safe constraint on root age, used if no fossil for root.11model = 0 * 0:JC69, 1:K80, 2:F81, 3:F84, 4:HKY8513alpha = 0 * alpha for gamma rates at sites14ncatG = 5 * No. categories in discrete gamma15cleandata = 0 * remove sites with ambiguity data (1:yes, 0:no)?17BDparas = 1 1 0 * birth, death, sampling19kappa_gamma = 6 2 * gamma prior for kappa20alpha_gamma = 1 1 * gamma prior for alpha21rgene_gamma = 2 2 * gamma prior for overall rates for genes23sigma2_gamma = 1 10 * gamma prior for sigma^2 (for clock=2 or 3)24finetune = 1: 0.1 0.1 0.1 0.01 .5 * auto (0 or 1) : times, musigma2, rates, mixing, paras, FossilErr26print = 128burnin = 2000029sampfreq = 230nsample = 100000
命令行输入以下命令执行。
11mcmctree mcmctree.ctl获得的结果文件如下:
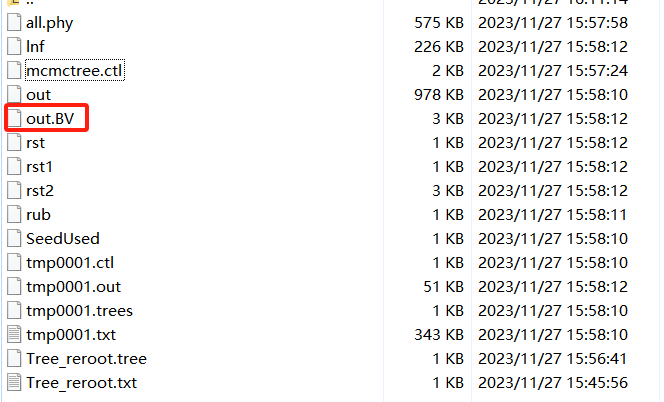
利用第一次运行结果中的out.BV作为输入文件,进行mcmctree的第二次建树。
新建Second_run_mcmctree.sh运行文件。
xxxxxxxxxx11091cd /home/hztext/dai/Comp_Genome/Nine_ortho_result/DiffTimetree2#新建第二次运行的目录3mkdir -p ../SecondRun4#拷贝第一次运行结果out.BV,重命名为in.BV5cp ./out.BV ../SecondRun/in.BV6#拷贝比对文件、树文件和mcmctree的参数文件7cp ./all.phy ../SecondRun/all.phy8cp ./Tree_reroot.tree ../SecondRun/Tree_reroot.tree9cp ./mcmctree.ctl ../SecondRun/mcmctree.ctl
准备mcmctree.ctl:(usedata设为2)
xxxxxxxxxx1311seed = -12seqfile = ./all.phy3treefile = ./Tree_reroot.tree4outfile = out5ndata = 1 ## 必须注意为 17seqtype = 2 * 0: nucleotides; 1:codons; 2:AAs ## 序列格式,蛋白就选择 AA8usedata = 2 * 0: no data; 1:seq like; 2:use in.BV; 3: out.BV ## 此步骤先设为 39clock = 3 * 1: global clock; 2: independent rates; 3: correlated rates10RootAge = <450.0 * safe constraint on root age, used if no fossil for root.11model = 0 * 0:JC69, 1:K80, 2:F81, 3:F84, 4:HKY8513alpha = 0 * alpha for gamma rates at sites14ncatG = 5 * No. categories in discrete gamma15cleandata = 0 * remove sites with ambiguity data (1:yes, 0:no)?17BDparas = 1 1 0 * birth, death, sampling19kappa_gamma = 6 2 * gamma prior for kappa20alpha_gamma = 1 1 * gamma prior for alpha21rgene_gamma = 2 2 * gamma prior for overall rates for genes23sigma2_gamma = 1 10 * gamma prior for sigma^2 (for clock=2 or 3)24finetune = 1: 0.1 0.1 0.1 0.01 .5 * auto (0 or 1) : times, musigma2, rates, mixing, paras, FossilErr26print = 128burnin = 2000029sampfreq = 230nsample = 100000
命令行输入以下命令执行。
11mcmctree mcmctree.ctl获得如下结果文件,FigTree.tre是我们需要的最终进化树文件。

4 FigTree软件美化物种进化树
FigTree下载链接:https://github.com/rambaut/figtree/releases
大家先自行摸索,后续再出美化教程哈哈哈哈哈哈哈哈!!!
